Chest X-Ray Medical Diagnosis with Deep Learning - ② Load the Datasets
start
BioinformaticsAndMe
Chest X-Ray Medical Diagnosis with Deep Learning
① Import Packages and Function
② Load the Datasets
③ Model Development
④ Training
⑤ Prediction and Evaluation
② Load the Datasets
: 환자 32,717명의 108,948개 frontal-view X-ray 이미지가 포함된 ChestX-ray8 데이터셋을 사용
→데이터셋의 각 이미지에는 14개의 병리학적 상태를 식별하는 여러 텍스트 마이닝 레이블이 포함됨
*의사는 이 결과를 통해, 8가지 질병을 진단하는데 사용할 수 있음
→이 데이터에 기반하여 14개의 레이블이 지정된 병리 상태에 대한 분류 모델을 개발할 것
*각 병리에 대해 '양성' 또는 '음성'을 예측
: Link 에서 전체 데이터셋을 다운로드할 수 있음
: 분석 실습은 축소된 아래 3개의 파일을 사용 (github.com/vishrutskaushik/Chest-X-Ray-Medical-Diagnosis)
- nih/train-small.csv: 875 images from our dataset to be used for training.
- nih/valid-small.csv: 109 images from our dataset to be used for validation.
- nih/test.csv: 420 images from our dataset to be used for testing.
: 이 데이터셋은 14개 병리 상태 중에서 아래 5개에 대해, 방사선과의사가 합의하여 주석을 달았음
- Consolidation
- Edema
- Effusion
- Cardiomegaly
- Atelectasis
#pandas 라이브러리로 데이터 로딩
#구글드라이브에 'nih' 폴더 업로드 후, 마운트
from google.colab import drive
drive.mount('/content/drive')
#데이터 로딩
train_df = pd.read_csv("drive/MyDrive/nih/train-small.csv")
valid_df = pd.read_csv("drive/MyDrive/nih/valid-small.csv")
test_df = pd.read_csv("drive/MyDrive/nih/test.csv")
#데이터 확인
train_df.head()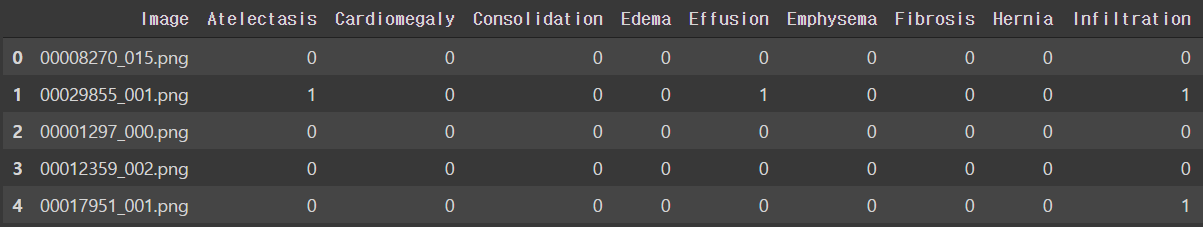
labels = ['Cardiomegaly',
'Emphysema',
'Effusion',
'Hernia',
'Infiltration',
'Mass',
'Nodule',
'Atelectasis',
'Pneumothorax',
'Pleural_Thickening',
'Pneumonia',
'Fibrosis',
'Edema',
'Consolidation']
2.1 Preventing Data Leakage
: 데이터셋에 각 환자에 대해 여러 이미지가 포함되어 있음
: 예를 들어, 환자가 병원 방문 중 여러 다른 시간에 X-ray 이미지를 촬영한 경우가 이에 해당
: Train, Validation,, Test 데이터셋 사이에 Leakage가 없도록, 데이터 분할이 수행됨
Exercise 1 - Checking Data Leakage
: 아래 셀에서 두 데이터셋 사이에 누출이 있는지 확인하는 함수가 작성됨
def check_for_leakage(df1, df2, patient_col):
"""
Return True if there any patients are in both df1 and df2.
Args:
df1 (dataframe): dataframe describing first dataset
df2 (dataframe): dataframe describing second dataset
patient_col (str): string name of column with patient IDs
Returns:
leakage (bool): True if there is leakage, otherwise False
"""
df1_patients_unique = set(df1[patient_col].unique().tolist())
df2_patients_unique = set(df2[patient_col].unique().tolist())
patients_in_both_groups = df1_patients_unique.intersection(df2_patients_unique)
# leakage contains true if there is patient overlap, otherwise false.
leakage = len(patients_in_both_groups) >= 1 # boolean (true if there is at least 1 patient in both groups)
return leakage: 예제 테스트
print("test case 1")
df1 = pd.DataFrame({'patient_id': [0, 1, 2]})
df2 = pd.DataFrame({'patient_id': [2, 3, 4]})
print("df1")
print(df1)
print("df2")
print(df2)
print(f"leakage output: {check_for_leakage(df1, df2, 'patient_id')}")
print("-------------------------------------")
print("test case 2")
df1 = pd.DataFrame({'patient_id': [0, 1, 2]})
df2 = pd.DataFrame({'patient_id': [3, 4, 5]})
print("df1:")
print(df1)
print("df2:")
print(df2)
print(f"leakage output: {check_for_leakage(df1, df2, 'patient_id')}"): 결과 출력
# Expected output
test case 1
df1
patient_id
0 0
1 1
2 2
df2
patient_id
0 2
1 3
2 4
leakage output: True
-------------------------------------
test case 2
df1:
patient_id
0 0
1 1
2 2
df2:
patient_id
0 3
1 4
2 5
leakage output: False: 다음 셀을 실행하여 train, valid, test 데이터셋에 공통된 환자가 있는지 확인
print("leakage between train and test: {}".format(check_for_leakage(train_df, test_df, 'PatientId')))
print("leakage between valid and test: {}".format(check_for_leakage(valid_df, test_df, 'PatientId')))
# Expected output
leakage between train and test: False
leakage between valid and test: False: 두 가지 모두에 대해 'False'가 출력되면, training을 위한 데이터셋 준비가 완료
2.2 Preparing Images
: 데이터셋 분할이 준비됐으므로, 이를 사용하도록 모델 설정을 진행할 수 있음
: 이를 위해, Keras의 ImageDataGenerator를 사용하여 데이터프레임에 지정된 이미지에 대한 'generator'를 빌드
: generator로 각 배치의 값을 변환하여 평균이 0, 표준 편차가 1이 되도록 표준화 함
: 또한, 단일채널 X-ray 이미지 (회색조)를 3채널 형식으로 변환
: 이미지 크기를 320x320 픽셀로 설정
def get_train_generator(df, image_dir, x_col, y_cols, shuffle=True, batch_size=8, seed=1, target_w = 320, target_h = 320):
"""
Return generator for training set, normalizing using batch
statistics.
Args:
train_df (dataframe): dataframe specifying training data.
image_dir (str): directory where image files are held.
x_col (str): name of column in df that holds filenames.
y_cols (list): list of strings that hold y labels for images.
sample_size (int): size of sample to use for normalization statistics.
batch_size (int): images per batch to be fed into model during training.
seed (int): random seed.
target_w (int): final width of input images.
target_h (int): final height of input images.
Returns:
train_generator (DataFrameIterator): iterator over training set
"""
print("getting train generator...")
# normalize images
image_generator = ImageDataGenerator(
samplewise_center=True,
samplewise_std_normalization= True)
# flow from directory with specified batch size
# and target image size
generator = image_generator.flow_from_dataframe(
dataframe=df,
directory=image_dir,
x_col=x_col,
y_col=y_cols,
class_mode="raw",
batch_size=batch_size,
shuffle=shuffle,
seed=seed,
target_size=(target_w,target_h))
return generator
Valid 및 Test 셋을 위한 별도의 생성기 구축
: 이제 데이터 validaiton 및 testing을 위한 새로운 생성기를 구축해야함
: Training 데이터와 동일한 generator를 사용할 수 없는 이유
-Training에서는 배치마다 각 이미지를 정규화하므로 배치 통계를 사용함
-실제 분석에서는 들어오는 이미지를 한 번에 일괄 처리하지 않음 (한번에 하나의 이미지를 처리)
-Training 모델에는 test 데이터셋에 대한 정보가 없어야 함
: 우리가 해야 할 일은 training 셋에서 계산된 통계를 사용하여, 들어오는 test 셋을 정규화하는 것
-아래 함수에서 이를 구현함
-이상적으로는 전체 training 셋을 사용하여 표본 평균과 표준 편차를 계산하고자 할 것
*그러나 이것은 매우 크기 때문에 시간이 많이 소요
-시간을 고려하여 데이터셋의 무작위 샘플을 취하고, 샘플 평균과 샘플 표준 편차를 계산함
def get_test_and_valid_generator(valid_df, test_df, train_df, image_dir, x_col, y_cols, sample_size=100, batch_size=8, seed=1, target_w = 320, target_h = 320):
"""
Return generator for validation set and test test set using
normalization statistics from training set.
Args:
valid_df (dataframe): dataframe specifying validation data.
test_df (dataframe): dataframe specifying test data.
train_df (dataframe): dataframe specifying training data.
image_dir (str): directory where image files are held.
x_col (str): name of column in df that holds filenames.
y_cols (list): list of strings that hold y labels for images.
sample_size (int): size of sample to use for normalization statistics.
batch_size (int): images per batch to be fed into model during training.
seed (int): random seed.
target_w (int): final width of input images.
target_h (int): final height of input images.
Returns:
test_generator (DataFrameIterator) and valid_generator: iterators over test set and validation set respectively
"""
print("getting train and valid generators...")
# get generator to sample dataset
raw_train_generator = ImageDataGenerator().flow_from_dataframe(
dataframe=train_df,
directory=IMAGE_DIR,
x_col="Image",
y_col=labels,
class_mode="raw",
batch_size=sample_size,
shuffle=True,
target_size=(target_w, target_h))
# get data sample
batch = raw_train_generator.next()
data_sample = batch[0]
# use sample to fit mean and std for test set generator
image_generator = ImageDataGenerator(
featurewise_center=True,
featurewise_std_normalization= True)
# fit generator to sample from training data
image_generator.fit(data_sample)
# get test generator
valid_generator = image_generator.flow_from_dataframe(
dataframe=valid_df,
directory=image_dir,
x_col=x_col,
y_col=y_cols,
class_mode="raw",
batch_size=batch_size,
shuffle=False,
seed=seed,
target_size=(target_w,target_h))
test_generator = image_generator.flow_from_dataframe(
dataframe=test_df,
directory=image_dir,
x_col=x_col,
y_col=y_cols,
class_mode="raw",
batch_size=batch_size,
shuffle=False,
seed=seed,
target_size=(target_w,target_h))
return valid_generator, test_generator: generator 함수가 준비 되었으면 training, validation, testing 데이터셋을 각각 하나씩 생성해봄
IMAGE_DIR = "./drive/MyDrive/nih/images-small/"
train_generator = get_train_generator(train_df, IMAGE_DIR, "Image", labels)
valid_generator, test_generator= get_test_and_valid_generator(valid_df, test_df, train_df, IMAGE_DIR, "Image", labels)# Expected output
getting train generator...
Found 1000 validated image filenames.
getting train and valid generators...
Found 1000 validated image filenames.
Found 200 validated image filenames.
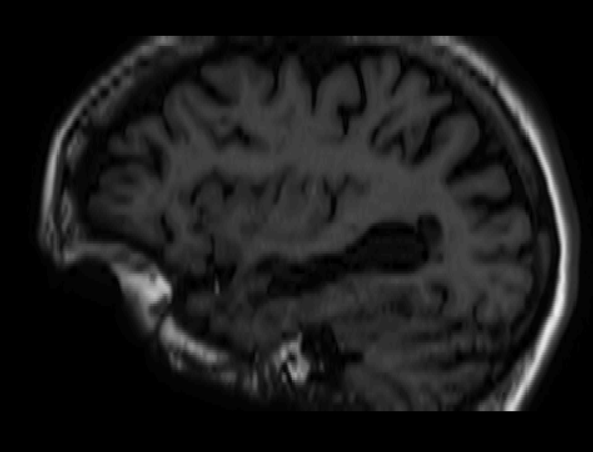
Found 420 validated image filenames.: '__get_item __ (index)' 함수를 호출하여, generator가 모델에 제공하는 내용을 살펴봄
x, y = train_generator.__getitem__(0)
plt.imshow(x[0]);
# Expected output
Clipping input data to the valid range for imshow with RGB data ([0..1] for floats or [0..255] for integers).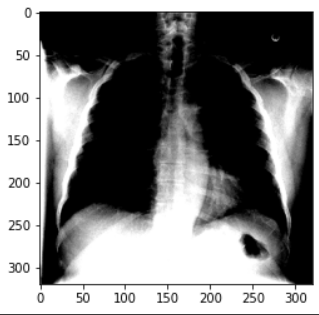
다음 분석
Chest X-Ray Medical Diagnosis with Deep Learning
③ Model Development
#Reference
1) www.coursera.org/learn/ai-for-medical-diagnosis
2) github.com/vishrutskaushik/Chest-X-Ray-Medical-Diagnosis
Chest X-Ray Medical Diagnosis with Deep Learning - ② Load the Datasets
end
BioinformaticsAndMe
'Machine Learning' 카테고리의 다른 글
| Chest X-Ray Medical Diagnosis with Deep Learning - ④ Training (0) | 2021.02.21 |
|---|---|
| Chest X-Ray Medical Diagnosis with Deep Learning - ③ Model Development (0) | 2021.02.21 |
| Chest X-Ray Medical Diagnosis with Deep Learning - ① Import Packages and Function (0) | 2021.02.15 |
| AUC-ROC 커브 (3) | 2020.08.04 |
| Support Vector Machine (SVM) (0) | 2020.02.05 |